Background
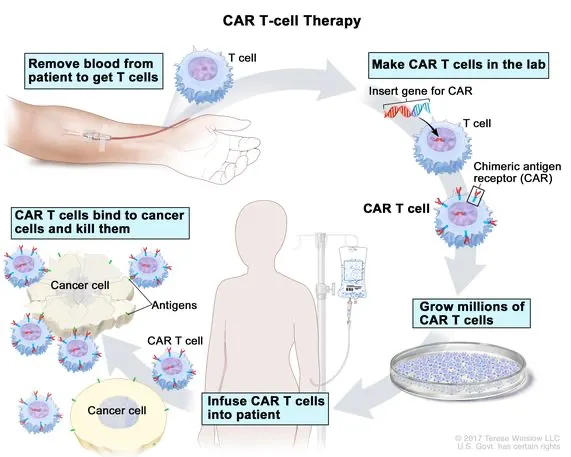
Chimeric antigen receptor (CAR) cell therapy
•
Cancer가 발현하는 antigen A를 알고 있다고 가정하자.
•
환자의 혈액에서 T세포를 추출해서, A에 대한 receptor를 발현하는 유전자를 넣어준다. 이렇게 만들어진 CAR-T 세포는 결과적으로 암세포만을 타겟하여 죽이는 능력을 얻게 된다.
•
체외에서 증식시켜서 다시 환자에게 넣어주면, 암세포를 죽인다.
Dual-target CAR engineering (CAR + Logical switch)
•
특정 AND, OR, NOT 조건을 만족할때만 killing이 일어나도록 어떻게 만듦?? 신기하다. 알아보자.
Motivation
CAR를 위한 좋은 Antigen을 고르는 방법?
•
뒤의 “Evaluation of CAR switch candidates in ovarian cancer” 파트의 결과가 motivation을 위한 좋은 예시가 된다.
•
예시)
◦
EPCAM은 OV에서 CAR cell therapy를 위한 antigen으로서 현재 활발하게 임상시험되고 있다.
◦
하지만 분석 결과 normal cell의 27.3%나 EPCAM을 발현할 만큼 off-tumor toxicity의 가능성이 있다. 위험함! → EPCAM을 어쨌거나 활용하고 싶은데, 어떻게 하면 좋을까?
◦
생각을 조금 확장해서, 그러면 EPCAM과 “암세포에서만” 함께 나타나는 antigen X를 찾고, EPCAM (AND) X 를 타겟하는 CAR를 디자인하면 어떨까?
▪
즉, EPCAM과 X가 함께 나타날 때만 세포를 죽임
◦
실제로 FOLR1는 암세포에서 EPCAM과 주로 함께 나타나지만, 정상 세포에서는 4.9%만 함께 나타남 → 보다 ideal한 candidate이라고 볼 수 있다!
•
A strategy to discover tumor antigens that are exclusively and homogeneously expressed in tumor tissues is of immediate urgency for successful CAR therapy.
•
암에서만 발현되는 antigen을 찾자!
•
Bulk 데이터로는 여러 세포 종류의 시그널이 섞여 있어서 근본적으로 어렵다 → Single cell 데이터를 쓰자!
◦
Cancer cell들을 어떻게 분리함? Cell type markers or aneuploidy status
◦
이러면 cancer cell에서만 exclusive하게 발현되는 antigen을 파악할 수 있고, 각 antigen 별로 얼마나 많은 cancer cell에서 발현을 보이는지를 계산함으로써 homogeneity를 판단할 수도 있다.
Targeting multiple antigens
•
to enhance anti-tumor responses and reduce the likelihood of immune escape in solid tumors
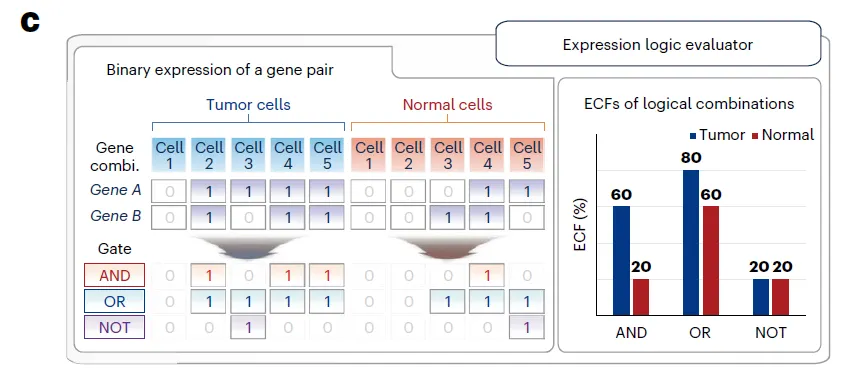
Logical gate를 이해하는 데 좋은 Figure
•
Dual-target CAR engineering → 각각의 CAR cell이 AND, OR, NOT 셋 중 하나의 logic gate로 기능하도록 설계
◦
AND = 하나의 cell이 두 antigen을 동시에 발현, OR = 하나의 cell이 두 antigen 중 하나만 발현, … 이런 개념인 것 같은데, 더 읽어보면서 이해해보자.
◦
개발이 덜 됐다고 주장. 기존 approach는 bulk RNA-seq 기반인데, 사실 antigen의 mutual expression pattern은 single cell RNA-seq 을 해야 정확히 알 수 있으므로
◦
예를 들어서, bulk 데이터는 AND (각각의 cell이 두 유전자를 동시에 발현)와 OR (두 그룹의 cell population이 두 유전자 중 하나를 발현) 로직을 구분할 수 없다.
◦
이런 expression logic은 암세포 말고도 정상 세포에서도 판단하는 것이 중요함.
▪
NOT-gated CARs require the AND expression of two antigens in normal cells for their protection
◦
“Expression logic”, “logical combination”, “logical switches”, “Boolean logic gates” 등등의 용어로 표현되고 있음.
이 논문에서는?
•
여러 Single-cell expression dataset을 효과적으로 통합하는 computational pipeline을 제시함.
◦
1,007,414 cells from 412 tumors of 17 types
◦
401,717 cells from 12 normal organs, encompassing 111 cell types
•
이 데이터셋에 Deep neural network에 기반한 알고리즘을 적용하여 surface antigen의 logical combination에 대한 meta-atlas를 구축함.
•
이 결과를 바탕으로 single cell 기반의 접근 방식이 optimized CAR logic design에 어떻게 기여할 수 있는지 논의함.
결과
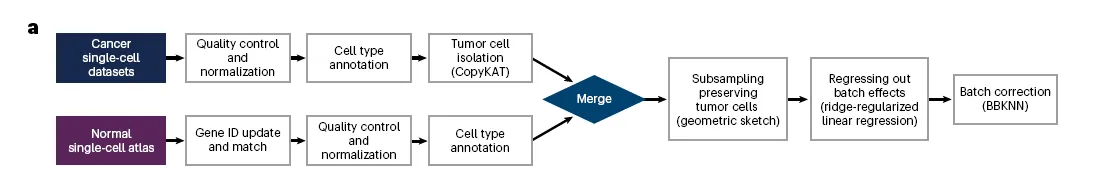
Construction of a tumor-normal single-cell meta-atlas
•
어떻게 데이터를 모으고 프로세싱해서 tumor-normal single-cell meta-atlas를 만들었는지 보자. 파이프라인은 아래와 같음.
Single-cell 데이터셋 합치기
•
Technical bias 줄이기 위해서 아래와 같은 조건의 데이터셋만 고름
◦
많이 쓰이는 파이프라인으로 생산된 데이터여야 함 (10x Genomics)
◦
Unscaled gene expression matrix 데이터여야 함 (count 말하는듯?)
◦
여러 donor가 포함된 데이터여야 함
•
Gene list를 통일하고, QC 잘 하고, (manual validation으로?) cell type annotation 함
•
Tumor 데이터의 경우 섞여있는 normal cell을 제거함
•
Geometric sketching 수행함
•
Batch correction 두번함
◦
한번은 Ridge regression으로 batch effect를 regress out 시키고
◦
이어서 Batch-balanced (BBKNN)으로 그래프 기반 batch correction
•
이렇게 single-cell meta-atlas가 만들어짐!
◦
1,007,414 cells from 412 tumors of 17 types
◦
401,717 cells from 12 normal organs encompassing 111 cell types
•
새로운 데이터가 생산되거나 새로 발견했을 때 atlas에 잘 합쳐질 수 있음을 보이기 위해서 난소암 샘플 9개 생산함.
•
잘 합쳐지는지 판단 어떻게?
1.
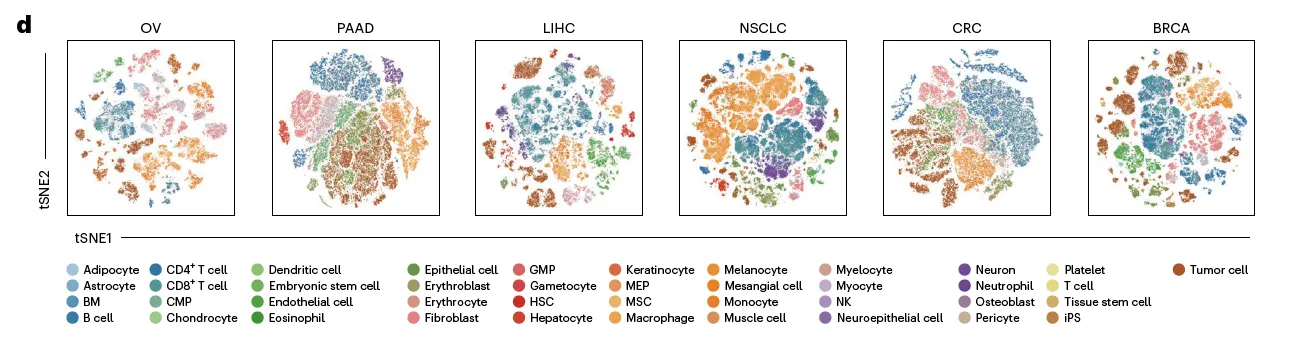
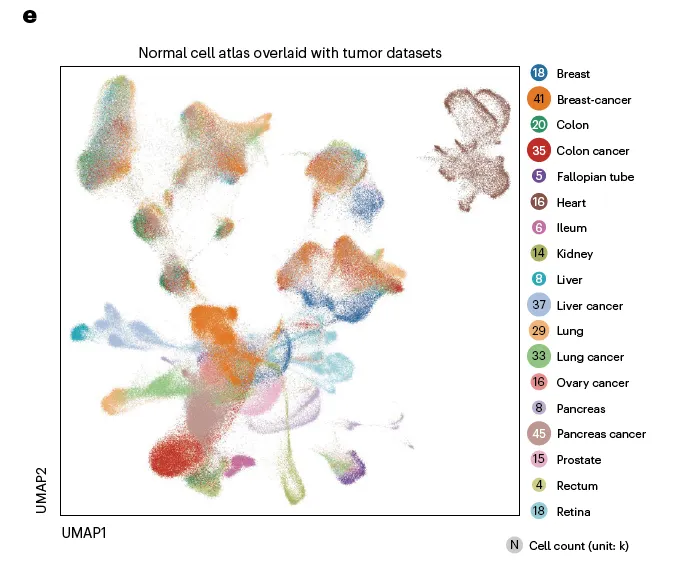
일단 6개 major tumor types (OV, NSCLC, CRC, BRCA, LIHC, PAAD)만 가지고 expression profile 시각화 해봄. (참고로 OV 데이터셋에 자체 생산된 9개 OV 샘플 포함해서 분석)
•
OV 포함 모든 암종에서 batch effect 없이 비슷한 cell들끼리 잘 뭉쳐짐을 확인할 수 있음.
2.
추가로 정상 세포들과 같이 찍었을 때 tissue of origin이 잘 반영된 그림이 나타나는지도 확인함.
•
잘 되더라.
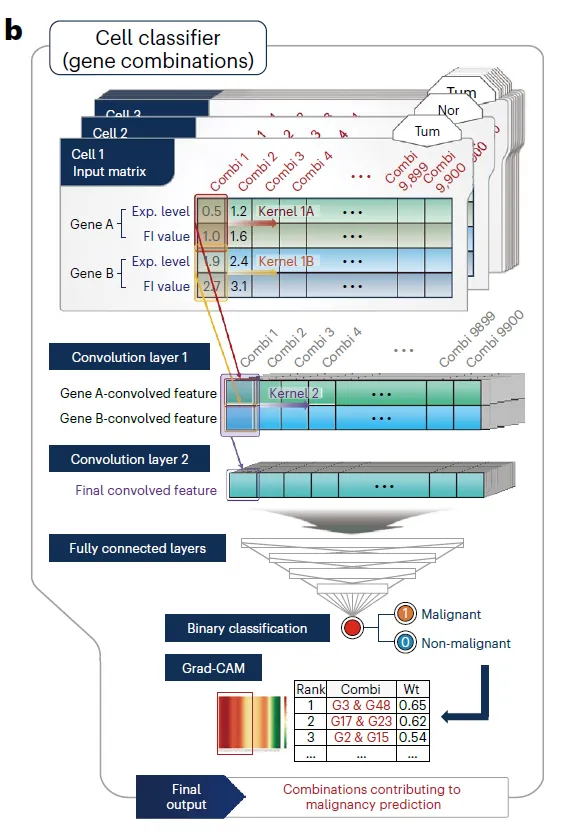
Optimal CAR target 찾기
•
결국 이 single-cell meta-atlas를 만든 목적은 optimal CAR target, 즉 암세포에서만 발현되는 유전자를 찾기 위함이다 (Surface면 더 좋겠지) → 이 유전자를 어떻게 찾았는지 보자
•
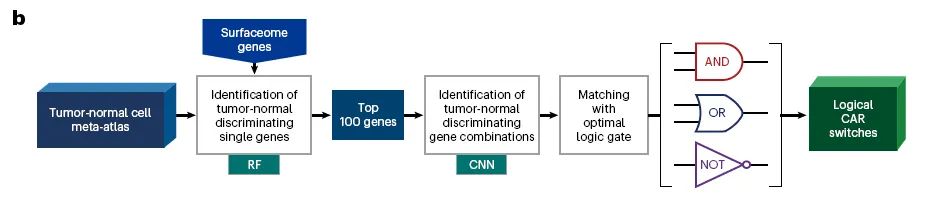
RF + CNN을 결합한 Two-step method로 malignant cell과 normal cell을 가장 잘 구분하도록 돕는 gene과 gene pair를 찾는 알고리즘 (feature selection 느낌)
•
이 알고리즘을 알려진 cancer surfaceome antigen 총 2,802개 유전자에 대해서 적용해봄.
•
RF module은 surfaceome antigen을 prioritize하려는 목적으로 디자인 되었고, CNN은 그 antigen들의 optimal logical combination을 알아내려는 목적으로 디자인되었다고 하는데, 어떻게 그게 가능한지는 좀 더 자세히 알아봐야겠음.
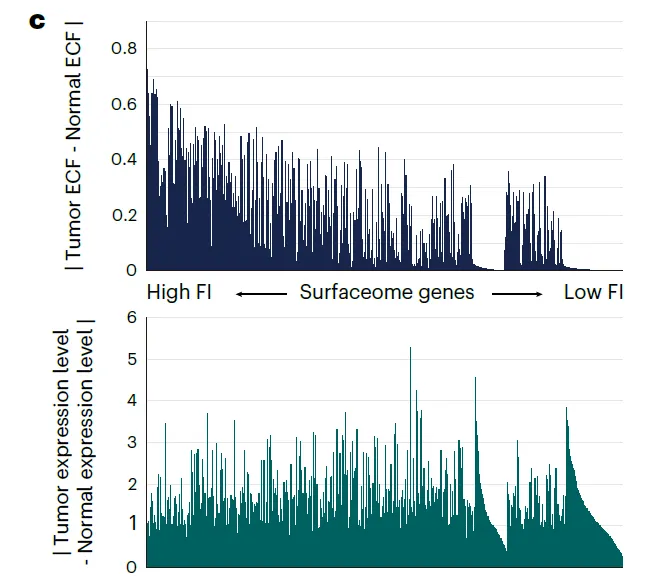
Importance of single-cell expression profiles
•
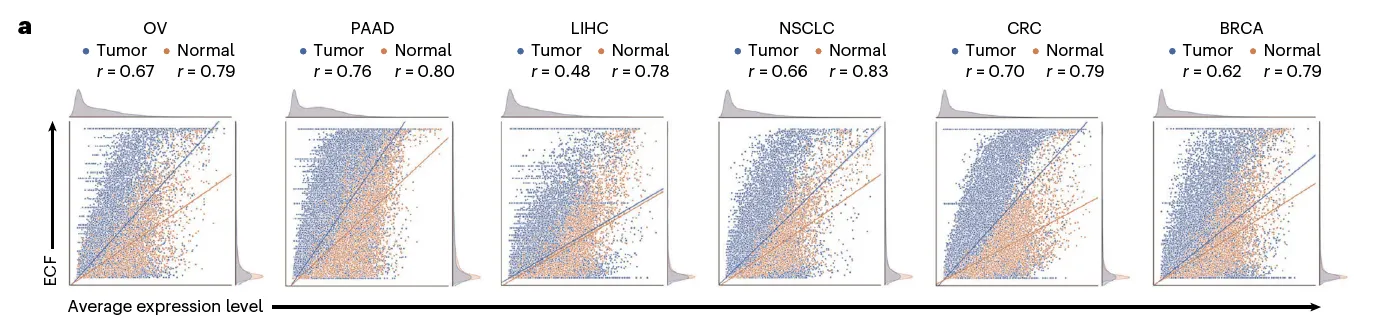
Expressing cell fraction (ECF) 라는 값을 정의: 주어진 유전자를 발현하고 있는 세포의 비율
•
6개 암종에 대해서 유전자의 average expression level과 ECF의 상관관계를 봄.
→ 해석?
•
전체적으로 correlation은 있지만, average expression level과 ECF 사이에 상당한 차이가 있었음 (?)
•
암 결과를 해석해보자.
◦
유전자들의 ECF가 암에서 특히 variable함 → 특정 clone에서만 발현되는 유전자가 있다는 것.
◦
결국 ECF가 높아야 (homogeneous하게 발현되어야) ITH가 적고, ideal한 candidate이라고 생각할 수 있음.
•
Normal 결과를 해석해보자.
◦
low ECF와 high expression에 biased 되어 있다.
◦
이런 유전자들 또한 ideal한 candidate이라고 볼 수 있는데, 그 이유는 적은 수의 normal cell 만이 영향을 받기 때문.
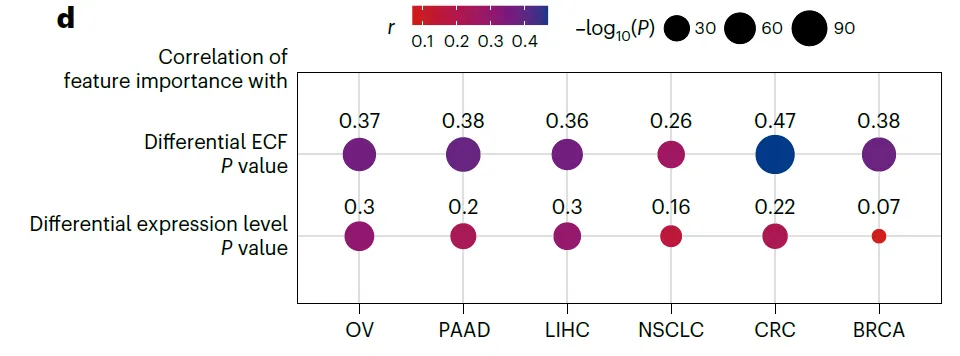
Identification of logical combinations for CAR switches
•
두 가지 모델을 사용함. Random forest (RF)와 Convolutional neural network (CNN)
•
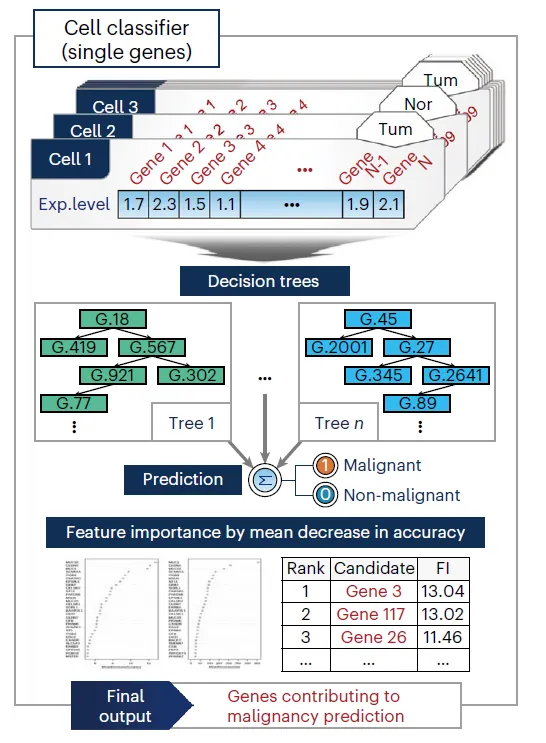
RF는 gene expression을 input으로 받아서 Tumor/Normal을 예측하는 모델
◦
→ Feature importance(FI)를 얻는다. Mean decrease in accuracy로 측정.
•
FI가 높게 잡힌 유전자들의 특징을 보면 Tumor/Normal에서의 ECF 차이는 크지만, expression level 차이는 딱히 크지 않다.
•
CNN은 “Gene pair”를 input으로 받아서 Tumor/Normal을 예측하는 모델
◦
Feature는 [Gene A의 발현량, Gene A의 FI, Gene B의 발현량, Gene B의 FI]
◦
Gene pair의, 모든 cell에 걸친 발현 패턴을 가지고 expression logic을 판단
Evaluation of CAR switch candidates in ovarian cancer
•
현재 OV에 대해 임상시험되고 있거나 bulk expression profiling으로 찾아낸 CAR switch candidate을 이 논문에서 제시하고 있는 방법으로 한번 평가해보자.
◦
과연 걔네들이 좋은 candidate일까? Logical combination으로 더 좋은 candidate을 찾을 수 있지 않을까?
•
95개의 previous target이 있었는데, tumor ECF가 50% 넘는 target이 7개밖에 안 되고, 심지어 그 target들은 normal ECF가 높더라 → 대부분이 암세포를 효과적으로 타겟하지 못하고, 타겟하더라도 off-tumor toxicity가 예상됨.
•
AND, OR, NOT gate를 활용하면 더 효과적이더라 (높은 tumor ECF, 낮은 normal ECF)
자세한 내용은…
•
모든 cell type 다 합친 ECF가 그렇게 크지 않다고 해도, cell-type 특이적인 ECF 값을 보면 특정 cell type에서 높게 나타날 수도 있다. 설계할 때 조심해야 함
•
비슷한 맥락에서, patient-specific한 스크리닝 과정이 필요할 수도 있음.
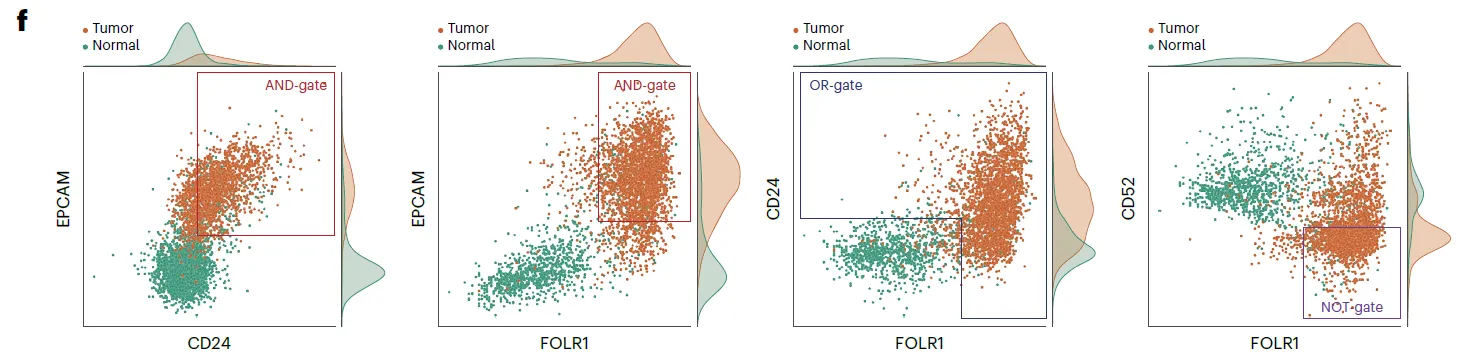
Single-cell epitope-based validation in ovarian cancer
•
여태까지는 transcriptome 수준의 logic을 가지고 분석한 것인데, 실제로 surface antigen으로 발현이 되어야 CAR를 디자인할 수도 있을 테니까 이를 검증함.
•
CITE-seq으로 검증함. CITE-seq은 single-cell 수준에서 surface protein과 transcriptome을 동시에 측정할 수 있는 기술.
•
자체 생산한 OV 샘플 3개를 pooling해서 사용함.
•
Validation targets (CITE-seq에 쓸 수 있는 Ab가 있어야 해서 필터링을 좀 함)
◦
EPCAM-and-CD24 (tumor ECF 90.2%)
◦
EPCAM-and-FOLR1 (82.0%)
◦
FOLR1-or-CD24 (97.2%)
◦
FOLR1-not-CD52 (81.0%)
•
CITE-seq → cell type annotation, tumor cell isolation을 했더니 2,541 tumor cell과 1,181 tumor-infiltrating normal cell을 얻음.
•
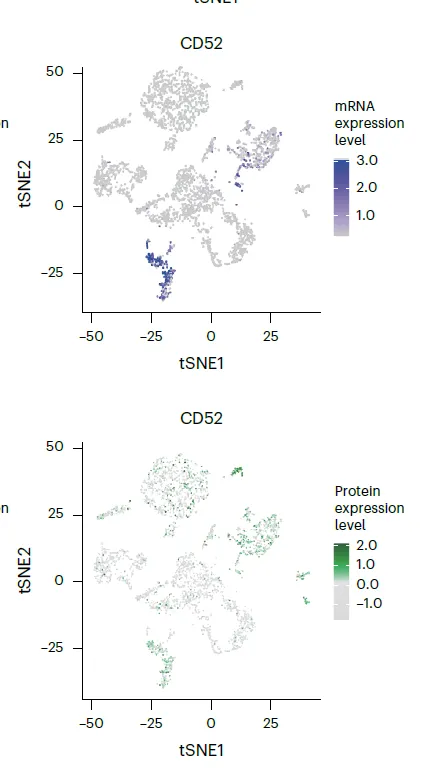
일단 cell 수준에서 mRNA 발현량과 surface protein 발현량이 좋은 상관관계를 보임.
◦
특히 CD52는 normal cell에서만 특이적으로 발현됨. → NOT gate에 써먹으면 유용
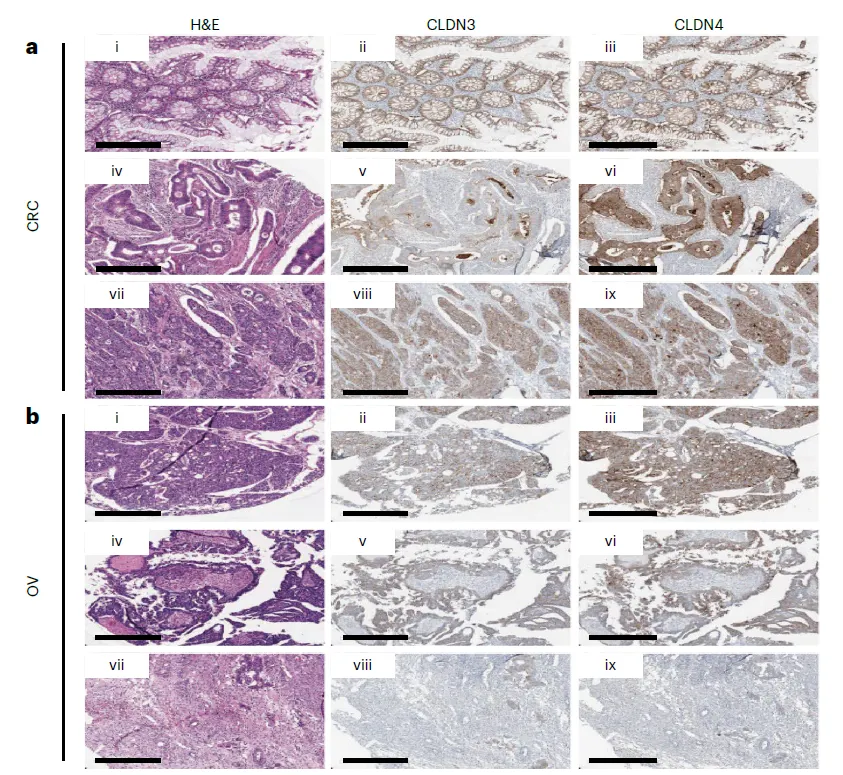
Protein-level validation in additional cancer types
•
CITE-seq으로 보는 것도 좋지만, experimental noise도 있고 antibody quality에 너무 의존한다는 단점이 있음. 그냥 IHC로 눈으로 함 보자
•
예를 들어서 CLDN3 / CLDN4 는 OV에서 ideal target으로 나타났는데 CITE-seq antibody는 작동을 잘 안 함. → IHC로 확인함